Como acessar dados públicos em R
Um guia prático para utilizar nosso datalake na linguagem R
TL;DR
Neste texto vamos explicar como usar a biblioteca basedosdados no R para explorar as diversas bases tratadas do datalake. Para ilustrar relação entre cobertura de saneamento básico e incidência de doenças de causa relacionada. Serão apresentadas as funções presentes do pacote e como utilizá-las para realizar análises.
Como acessar o datalake público
Organizamos no datalake as principais bases de dados públicas já tratadas e prontas para análise. O datalake é mantido no ambiente da Google (BigQuery) e o acesso às bases é gratuito, com um limite mensal de 1TB por mês — acredite, nem a gente chega a tanto.
O pacote basedosdados te permite acessar esse banco através do R de um jeito rápido e fácil. Para isso, é necessário que você possua um projeto (gratuito) no Google Cloud — veja como criar seu projeto com estes 5 passos ou siga as instruções na primeira vez que usar o pacote.
Conhecendo a biblioteca basedosdados
Como qualquer outra biblioteca no R, você deve instalá-la e carregar no seu ambiente:
# instalando a biblioteca
install.packages("basedosdados")
# carregando a biblioteca na sessão
library(basedosdados)
A biblioteca contém duas funções principais:
download(), que permite baixar bases do datalake como arquivo .CSVread_sql(), que já abre uma base em formato tibble na sua sessão R.
Função download()
Para utilizar a função download() , você pode usar os argumentos:
query: query em SQL com qual tabela se quer baixar, com quais colunas e com qual agregaçãopath: um caminho para onde salvar o arquivo.csv.
Um truque é usar projetos do R. Com eles, você não precisa especificar todo o caminho onde quer salvar as bases e o código pode ser reutilizado por outros usuários. Ao longo do texto vamos usar o caminho
/basescomo uma pasta em um dado projeto R. Note que depois de/basesacrescentamos o nome do arquivo e o formato .csv.
Para baixar os dados do Atlas Esgotos da ANA, você pode rodar:
basedosdados::download(
query = 'SELECT * FROM `basedosdados.br_ana_atlas_esgotos.municipio`',
path = '/bases/base_ana.csv'
)
Função read_sql()
A read_sql() usa uma API para acessar o datalake e abrir uma base em formato tibble na sua sessão de R. O R se encarrega de entrar na nuvem, escolher a tabela e rodar a query para acessá-la, conforme os parâmetros passados na função:
query(string) : é a consulta que será feita ao banco em SQL. Aqui você pode especificar filtros, agrupamentos, e outras transformações usando SQL. Veja mais sobre a sintaxe aqui.billing-project-id: é o identificador do seu projeto do Google. Ele é necessário para que o Google saiba quem está acessando a nuvem, e contabilizar o tamanho da requisição que você está fazendo. Não precisa ser especificado caso aset_billing_idseja usada.
Funções set_billing_id() e get_billing_id()
A set_billing_id() guarda o seu billing-project-id usado na função read_sql(). Em outras palavras, você pode definir uma única vez na sessão o seu identificador de projeto do Google, e ele não precisa mais ser um argumento para acessar tabelas. Para os mais experientes, um outro truque possível é guardar o seu id em algum objeto de um arquivo .env e depois usar a função Sys.getenv(objecto) para acessá-lo. Um arquivo .env pode ser gerado com a biblioteca dotenv.
A get_billing_id() printa o valor guardado em set_billing_id. Essa função não é diretamente usada: a função read_sql() automaticamente pega o identificador definido através da get_billing_id().
Dito isso, uma vez que definimos nosso billing-project-id:
basedosdados::set_billing_id("meu-projeto-3058")
Não precisamos mais usar a função read_sql() com dois argumentos:
basedosdados::read_sql(
query = "SELECT * FROM `basedosdados.br_ana_atlas_esgotos.municipio`",
billing_project_id = "meu-projetoid-3058"
)
Podemos usá-la sem o segundo argumento (que é o que será feito ao longo do texto):
basedosdados::read_sql(
query = "SELECT * FROM `basedosdados.br_ana_atlas_esgotos.municipio`"
)
Aplicações
Antes: Como descobrir os nomes de tabelas
Para exemplificar o uso da biblioteca, vamos estudar duas variáveis de bases diferentes que acreditamos estar correlacionadas: investimento em coleta de esgoto e mortes por doenças que se propagam na falta de saneamento básico.
Para começar essa análise, iremos acessar os dados do Atlas Esgotos da Agência Nacional das Águas. Ao acessar o link do conjunto de dados no nosso site (acima), você verá listado as “Tabelas tratadas” — isso indica quais tabelas estão disponíveis no banco.
Neste caso temos somente a tabela municipios. Ao clicar na mesma, você encontra a seção “Consulta aos Dados”, que mostra como acessar essa tabela no próprio banco (via editor do BigQuery), em Python e em R (figura abaixo).
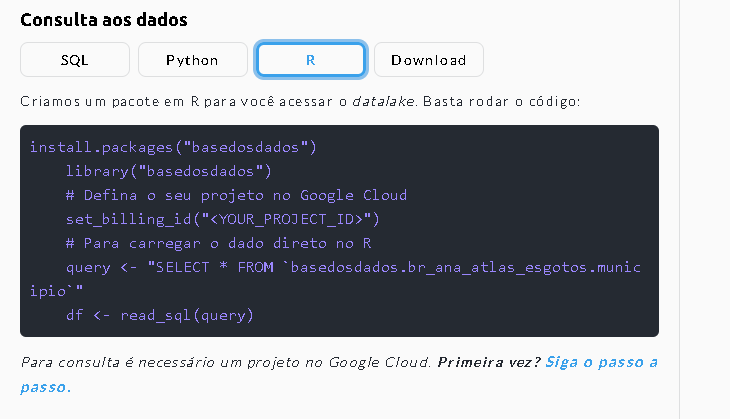
No código, vem descrito já o nome do conjunto e da tabela no banco — neste caso, basedodados é o banco, br_ana_atlas_esgotos o conjunto e municipios a tabela. Você pode copiar este código direto para o RStudio.
Diferentes maneiras de consultar uma tabela no R
Utilizando a função read_sql(), iremos carregar os dados do Atlas Esgostos da ANA no R e salvar esse tibble em um objeto chamado base.
base <- basedosdados::read_sql(
query = "SELECT * FROM `basedosdados.br_ana_atlas_esgotos.municipio`"
)
Caso você clique para ver a base, vai se deparar com algo assim:
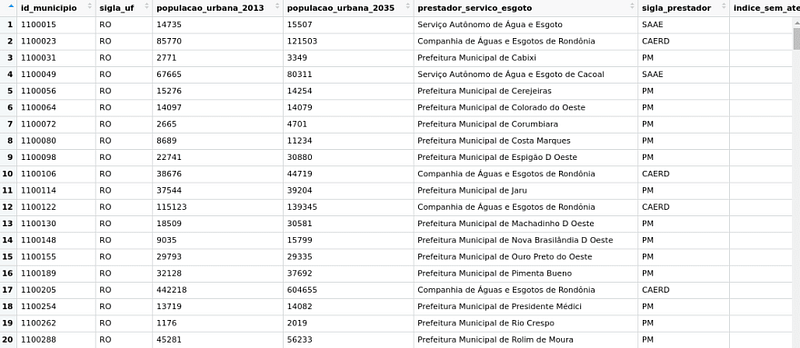
O Atlas contém mais de 30 variáveis sobre a condição da coleta e do tratamento de esgoto para cada município brasileiro. Entre elas, temos, por exemplo, a porcentagem de habitantes do município sem acesso a esgoto tratado, o nome do prestador do serviço do saneamento e o investimento feito pelo município em coleta e em tratamento de esgoto.
A query usada contém um * para indicar que estamos selecionando todas as colunas da tabela. Caso quiséssemos baixar só duas colunas, como o identificador do municipio (id_municipio) e o índice de pessoas que não recebe atendimento de tratamento de esgoto (indice_sem_atendimento_sem_coleta_sem_tratamento) bastaria rodar algo como:
base_cobertura <- basedosdados::read_sql(
query = "
SELECT
id_municipio,
indice_sem_atendimento_sem_coleta_sem_tratamento
FROM
`basedosdados.br_ana_atlas_esgotos.municipio`
"
)
Outra maneira de selecionar uma "sub-base" é filtrando as observações por alguma característica: se estivermos interessados somente no saneamento básico da região norte, não faz sentido pegarmos todas as 5570 linhas da base original. Podemos rodar uma query adicionando o verbo WHERE e indicar que só queremos estados do Norte:
base_norte <- basedosdados::read_sql('
SELECT
*
FROM
`basedosdados.br_ana_atlas_esgotos.municipio`
WHERE
sigla_uf IN ("AM",
"AP",
"RO",
"RR",
"AC",
"PA")
')
Além dessas possibilidades de seleção, podemos agregar essas tabela para o nível de estado, ao invés do nível de município, utilizando um agrupamento por sigla_uf. Ao agregarmos, precisamos também agregar as colunas, somando ou tirando uma média, por exemplo. Nesse exemplo, vamos pegar uma média da cobertura de esgoto por UF. O código fica assim:
base_uf <- basedosdados::read_sql(query = '
SELECT
sigla_uf,
AVG(indice_sem_atendimento_sem_coleta_sem_tratamento) AS sem_esgoto
FROM `basedosdados.br_ana_atlas_esgotos.municipio`
GROUP BY
sigla_uf
')
Visualizando o acesso a tratamento nos estados
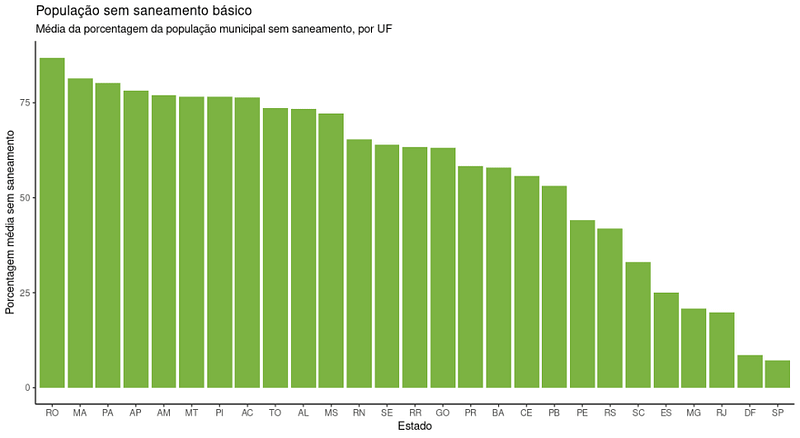
Para finalizar, vamos construir um gráfico com essa última base criada para visualizar quais estados tem maior e a menor média de acesso a tratamento de esgoto nos seus municipios:
base_uf %>%
ggplot(aes(y = sem_esgoto, x = reorder(sigla_uf, -sem_esgoto))) +
geom_col(fill = "#7cb342") +
labs(
x = "Estado",
y = "Porcentagem média sem saneamento",
title = "População sem saneamento básico",
subtitle = "Média da porcentagem da população municipal sem saneamento, por UF"
) +
theme_classic()
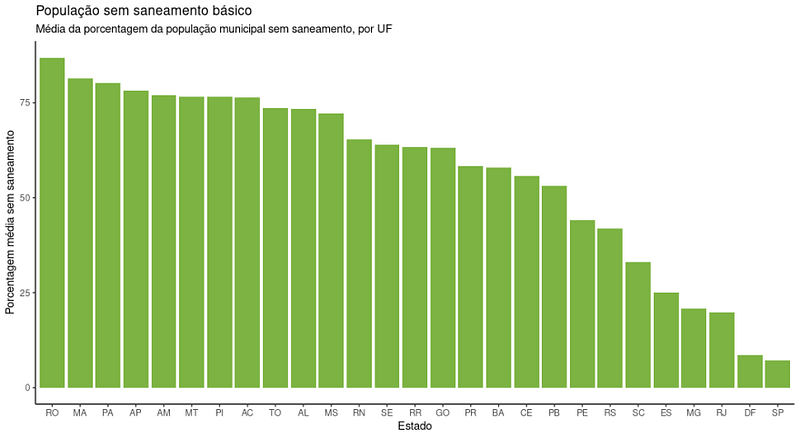
Parabéns! Concluímos a nossa primeira aplicação da basedosdados! Com ela, descobrimos que o estado que tem, em média, o pior acesso a tratamento e coleta de esgoto nas suas cidades é Rondônia, e o estado que tem a melhor é São Paulo.
Para usar esta mesma base em um outro software, pode-se usar a função download() para baixá-la em CSV direto na sua máquina:
basedosdados::download(
query = "
SELECT
sigla_uf,
AVG(indice_sem_atendimento_sem_coleta_sem_tratamento) AS sem_esgoto
FROM
`basedosdados.br_ana_atlas_esgotos.municipio`
GROUP BY
sigla_uf
",
path = "/bases/base_ana_uf.csv"
)
Cruzando diferentes bases
Outra aplicação importante do pacote é a possibilidade de juntar diferentes bases sem ter que abrí-las individualmente.
Para exemplificar, vamos comparar os dados que obtemos de saneamento com o nível de mortalidade por doenças relacionadas à falta de saneamento. Para explorar mortalidade precisamos de número de óbitos, que estão na tabela do Sistema de Mortalidade do Ministério da Saúde (SIM), e da população, na tabela de população do IBGE. Ambas as tabelas estão disponíveis na BD nos links acima!
Para cruzar as tabelas vamos filtrar ambas para o ano de 2013, referente ao Atlas Esgotos (tabela anterior), pela coluna ano presente em todas as tabelas. Além disso, vamos também escolher somente a mortalidade de causa_basica referente a doenças diarréicas, relacionadas à falta de saneamento básico. Os códigos de referência da coluna causa_basica na tabela SIM podem ser consultados aqui. A query abaixo faz esses filtros e seleciona as colunas tanto da base de população e quanto de mortalidade:
base_mortalidade <- basedosdados::read_sql('
SELECT
sim.id_municipio,
sim.numero_obitos,
pop.populacao
FROM
`basedosdados.br_ms_sim.municipio_causa` AS sim
FULL JOIN
`basedosdados.br_ibge_populacao.municipio` AS pop
ON
sim.id_municipio = pop.id_municipio
WHERE
sim.ano = 2013
AND pop.ano = 2013
AND sim.causa_basica IN ("A00",
"A01",
"A02",
"A03",
"A04",
"A05",
"A06",
"A07",
"A08",
"A09")
'
) %>%
mutate(mortalidade = (numero_obitos / populacao) * 10000) %>%
select(id_municipio, mortalidade)
Vamos então juntar essa base com a tabela de cobertura de saneamento e ver as possíveis correlações. Para isso, podemos juntar as bases abertas no R, a base_cobertura e a base_mortalidade, ou podemos rodar uma nova QUERY:
base_final <- basedosdados::read_sql('
SELECT
sim.id_municipio,
sim.numero_obitos,
pop.populacao,
ana.indice_sem_atendimento_sem_coleta_sem_tratamento
FROM
`basedosdados.br_ms_sim.municipio_causa` AS sim
FULL JOIN
`basedosdados.br_ibge_populacao.municipio` AS pop
ON
sim.id_municipio = pop.id_municipio
FULL JOIN
`basedosdados.br_ana_atlas_esgotos.municipio` AS ana
ON
sim.id_municipio = ana.id_municipio
WHERE
sim.ano = 2013
AND pop.ano = 2013
AND sim.causa_basica IN ("A00",
"A01",
"A02",
"A03",
"A04",
"A05",
"A06",
"A07",
"A08",
"A09")
'
)
Com a base em mãos, criamos um gráfico que relaciona a mortalidade por doenças diarreicas e a cobertura de saneamento básico:
base_final %>%
mutate(mortalidade = (numero_obitos / populacao) * 10000) %>%
ggplot(aes(y = mortalidade, x = indice_sem_atend)) +
geom_point(color = "#7cb342") +
labs(
x = "Porcentagem sem saneamento",
y = "Mortalidade",
title = "Saneamento x Mortalidade",
subtitle = "Os municípios que tem pior cobertura também tem mais mortes?"
) +
theme_classic()
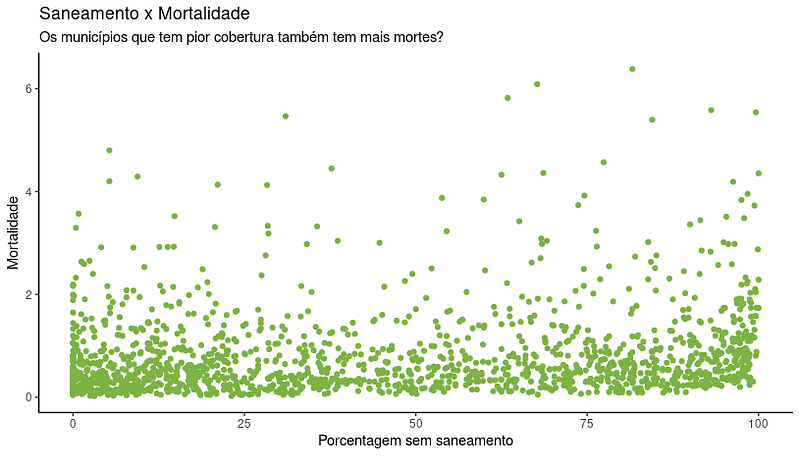
E com isso descobrimos que aparentemente não há uma forte correlação entre cobertura de saneamento e mortalidade por doenças quando analisamos todos municípios brasileiros que tiveram mortes por diarreia… Mas, conseguimos facilmente juntar 3 bases de diferentes organizações numa única entrada!
Muito obrigado por ler até aqui!
Confira o notebook com a análise completa no nosso repositório.
Notou algo errado ou tem uma sugestão?
Contribua com a BD editando este artigo via pull request no nosso GitHub.Base dos Dados