Analisando dados de vacinação contra COVID-19 com a BD
Veja como utilizar o datalake público da Base dos Dados para criar um gráfico de vacinação da sua cidade
TL;DR
Uma das melhores formas de comunicar grandes quantidades de dados é por meio de gráficos. Hoje vamos aprender como buscar os dados da [Campanha Nacional de Vacinação contra Covid-19](http://Campanha Nacional de Vacinação contra Covid-19) e de população na Base dos Dados, além de como gerar um gráfico de vacinados de acordo com a população, utilizando apenas o pacote em R da basedosdados e o ggplot2.
Introdução e contexto
Desde o início da pandemia, fomos inundados com números de casos, óbitos, ocupação hospitalar e, mais recentemente, de vacinação! Com essa avalanche de números diários ficou bem clara a importância dos gráficos para comunicação. Como biólogo, adoro pensar em como nós, seres humanos, somos organismos visuais e evoluímos por milhares de anos buscando padrões na natureza que nos possibilitassem sobreviver. Nosso cérebro adora desenhos, gráficos e figuras, por isso devemos usá-los para comunicar dados!
Foi com essa ideia na cabeça que comecei a fazer gráficos relacionados à COVID-19 para minha cidade, Maringá-PR. E, claro, o gráfico mais legal de fazer é o da vacinação. O brasileiro ama se vacinar!
Mas não foi tão fácil assim buscar esses dados. Entrar no site do SUS, baixar os microdados de vacinação do estado (que aumentava a cada semana), filtrar apenas da minha cidade (precisava usar o Kaggle pois o R travava!), exportar uma tabela, para só então eu fazer os gráficos. Atualizar diariamente esse gráfico era impossível!
Era, até que conheci a Base dos Dados! Buscar bancos de dados públicos dentro do R ficou tão fácil que parecia mentira e hoje vou compartilhar um pouco sobre como você pode fazer um gráfico da vacinação de acordo com o sexo e faixa etária, comparando com a população estimada em sua cidade.
Pacotes utilizados
Vamos carregar os poucos pacotes que utilizados para essa visualização
library(basedosdados) # buscar os dados
library(tidyverse) # manipulação dos dados
library(extrafont) # utilizar fontes do nosso computador caso queira
Busca dos dados
Essa parte seria a mais trabalhosa, mas agora é a mais simples. Utilizando o pacote em R da basedosdados, só precisamos rodar:
# buscar os dados de vacinação
dados_vacina <- bdplyr("basedosdados.br_ms_vacinacao_covid19.microdados") %>%
# vamos filtrar o estado e o município pelo código do IBGE
filter(
sigla_uf == "PR",
id_municipio_estabelecimento == "4115200"
) %>%
# vamos selecionar apenas as variáveis a serem utilizadas
select(
data_aplicacao_vacina,
idade_paciente,
sexo_paciente,
dose_vacina
) %>%
# agora é só buscar!
bd_collect()
# buscar os dados da população em 2020
populacao_mga <- bdplyr("basedosdados.br_ms_populacao.municipio") %>%
# vamos filtrar o município pelo código do IBGE e o ano de 2020
filter(
id_municipio == "4115200",
ano == 2020
) %>%
# vamos selecionar apenas as variáveis a serem utilizadas
select(
sexo,
grupo_idade,
populacao
) %>%
# vamos buscar!
bd_collect()
Pronto! Já temos o dataset com os microdados dos vacinados (dados_vacina) e a estimativa da população (populacao_mga) carregados no R!
Nesse texto utilizei a minha cidade como exemplo (código 4115200), mas você pode fazer para a sua também. É só buscar sua cidade no site IBGE Cidades, colocar o código do IBGE de 7 dígitos nos filtros de id_municipio_estabelecimento e id_municipio acima e mudar a sigla_uf para seu estado!
Arrumando os dados
Agora que já temos os dados prontinhos, só precisamos organizar alguns pontos para fazermos nosso gráfico!
Dados da vacinação
Vamos retirar as doses de reforço (já que estamos focando apenas na 1ª e 2ª dose) e quando o sexo é “indeterminado” (sim, os dados possuem alguns erros). Além disso, como os dados de dose estão escritos como 1ª Dose e 2ª dose, vamos mudar para 1 e 2.
dados_vacina2 <- dados_vacina %>%
rename(sexo = "sexo_paciente") %>%
filter(
dose_vacina != "Reforço",
sexo != "I"
) %>%
mutate(dose_vacina = ifelse(str_detect(dose_vacina, "1"), 1, 2))
Aproveitando o gancho do "sexo indeterminado", precisamos entender que os dados públicos - principalmente estes que são alimentados por diferentes pessoas de diferentes prefeituras de todo o país - sempre estarão passivas de erros. Álvaro, da brasil.io fez um post muito bom com alguns pontos acerca dos dados de vacinação.
Se temos erros, o que podemos fazer? Na medida do possível, desconsideraremos esses erros (como fizemos acima), lembrando que são poucos dados com erros grosseiros. Mesmo com esses problemas, essa é nossa fonte de dados mais confiável para essa análise!
Continuando com os dados, vamos agrupar os vacinados por categorias de 5 em 5 anos, assim como os dados que encontramos na projeção do Ministério da Saúde (populacao_mga), além de contar quantas pessoas existem em cada grupo de idade, sexo e dose recebida!
dados_vacina3 <- dados_vacina2 %>%
mutate(grupo_idade = case_when(
idade_paciente <= 4 ~ "0-4 anos",
idade_paciente >= 5 & idade_paciente <= 9 ~ "5-9 anos",
idade_paciente >= 10 & idade_paciente <= 14 ~ "10-14 anos",
idade_paciente >= 15 & idade_paciente <= 19 ~ "15-19 anos",
idade_paciente >= 20 & idade_paciente <= 24 ~ "20-24 anos",
idade_paciente >= 25 & idade_paciente <= 29 ~ "25-29 anos",
idade_paciente >= 30 & idade_paciente <= 34 ~ "30-34 anos",
idade_paciente >= 35 & idade_paciente <= 39 ~ "35-39 anos",
idade_paciente >= 40 & idade_paciente <= 44 ~ "40-44 anos",
idade_paciente >= 45 & idade_paciente <= 49 ~ "45-49 anos",
idade_paciente >= 50 & idade_paciente <= 54 ~ "50-54 anos",
idade_paciente >= 55 & idade_paciente <= 59 ~ "55-59 anos",
idade_paciente >= 60 & idade_paciente <= 64 ~ "60-64 anos",
idade_paciente >= 65 & idade_paciente <= 69 ~ "65-69 anos",
idade_paciente >= 70 & idade_paciente <= 74 ~ "70-74 anos",
idade_paciente >= 75 & idade_paciente <= 79 ~ "75-79 anos",
idade_paciente >= 80 ~ "80_mais"
)) %>%
select(grupo_idade, sexo, dose_vacina) %>%
group_by(grupo_idade, sexo, dose_vacina) %>%
summarise(n = dplyr::n())
Dados da população
A estimativa da população feita pelo Ministério da Saúde serve para que o governo tenha uma ideia do provável número de pacientes do SUS, ou mesmo quantas doses devem ser enviadas para cada município. Essa estimativa é feita com base no último Censo (de 2010) e, justamente por ter sido feito há bastante tempo, a estimativa não será tão precisa(olha aí mais uma importância do Censo). Mas, como vimos anteriormente, é o melhor que temos. Aqui precisamos apenas igualar os dados com a nossa tabela de vacinados.
populacao_mga <- populacao_mga %>%
mutate(sexo = recode(sexo,
"masculino" = "M",
"feminino" = "F"
))
Combinando dados de Vacinação e População
O código abaixo mostra como combinar os dados de vacinação com os de população:
vacina_populacao <- dados_vacina3 %>%
left_join(populacao_mga, by = c("grupo_idade", "sexo"))
Produção do gráfico
O gráfico que queremos fazer tem mais ou menos a estrutura da imagem 1. No entanto, com o ggplot2 é mais fácil fazermos um gráfico de colunas, como a imagem 2, e depois apenas inverter os eixos x e y utilizando a função coord_flip() no final da produção do gráfico.
Por esse motivo, toda a construção do nosso gráfico será feita com base no gráfico 2 acima. Assim, os números referentes ao sexo masculino (em laranja) serão tratados como negativos, enquanto os do sexo feminino (verde-água?) serão tratados como positivos. É o que faremos no script a seguir, além de adicionar a porcentagem de vacinados para cada grupo.
vacina_populacao2 <- vacina_populacao %>%
mutate(
n_grafico = ifelse(sexo == "M", n * (-1), n),
populacao_grafico = ifelse(sexo == "M", populacao * (-1), populacao),
porc = round((n / populacao) * 100, digits = 1)
)
Como um toque final em nossos dados, vamos apenas determinar a ordem que queremos que os grupos etários sejam exibidos no gráfico 0–4 anos -> 80_mais
vacina_populacao2$grupo_idade <- factor(vacina_populacao2$grupo_idade,
levels = c(
"0-4 anos",
"5-9 anos",
"10-14 anos",
"15-19 anos",
"20-24 anos",
"25-29 anos",
"30-34 anos",
"35-39 anos",
"40-44 anos",
"45-49 anos",
"50-54 anos",
"55-59 anos",
"60-64 anos",
"65-69 anos",
"70-74 anos",
"75-79 anos",
"80_mais"
)
)
Além disso, vamos também determinar quais cores queremos para cada categoria. Vale lembrar que é muito importante não reafirmar o estereótipo de cores para gêneros. Um texto muito legal sobre isso foi escrito no blog do Datawrapper. Por isso, vamos utilizar #EE5A45 como base para o sexo masculino e #1E8F89 como base para o sexo feminino.
cores <- c(
"População estimada (Masculino)" = "#ffc3a4",
"1ª Dose (M)" = "#EE5A45",
"2ª Dose ou Única (M)" = "#790000",
"População estimada (Feminino)" = "#7bd1cb",
"1ª Dose (F)" = "#1E8F89",
"2ª Dose ou Única (F)" = "#00322f"
)
Agora vamos fazer o gráfico! Eu trouxe para vocês 2 tipos de gráficos, um mais simples e outro mais completo, com diversos detalhes.
Gráfico simples
Com esse código abaixo, você pode fazer e entender rapidamente o gráfico da sua cidade! Retirei diversos detalhes e parâmetros específicos para a população da minha cidade. Podemos fazer um gráfico bem simples, sem muitos detalhes:
# definir o dataset e os eixos (invertidos)
ggplot(vacina_populacao2, aes(x = grupo_idade, y = n_grafico)) +
# populacao estimada
## feminino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
distinct(populacao_grafico, .keep_all = TRUE),
aes(
x = grupo_idade,
y = populacao_grafico,
fill = "População estimada (Feminino)"
),
stat = "identity"
) +
## masculino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
distinct(populacao_grafico, .keep_all = TRUE),
aes(
x = grupo_idade,
y = populacao_grafico,
fill = "População estimada (Masculino)"
),
stat = "identity"
) +
# 1_dose
## feminino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
filter(dose_vacina == "1"),
aes(fill = "1ª Dose (F)"),
stat = "identity"
) +
## masculino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
filter(dose_vacina == "1"),
aes(fill = "1ª Dose (M)"),
stat = "identity"
) +
# 2_dose
## feminino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
filter(dose_vacina == "2"),
aes(fill = "2ª Dose ou Única (F)"),
stat = "identity"
) +
## masculino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
filter(dose_vacina == "2"),
aes(fill = "2ª Dose ou Única (M)"),
stat = "identity"
) +
# eixos
labs(
x = "Faixa etária",
y = "Vacinados",
title = "Vacinação em Maringá-PR",
subtitle = "Vacinados por faixa etária até 19/11/2021",
caption = "Gráfico: Bruno H. Mioto Stabile - @BrunoHMioto - Fonte: basedosdados.org (Vacinação COVID-19 e População)"
) +
# definir cores
scale_fill_manual(
values = cores,
limits = c(
"População estimada (Masculino)",
"1ª Dose (M)",
"2ª Dose ou Única (M)",
"2ª Dose ou Única (F)",
"1ª Dose (F)",
"População estimada (Feminino)"
)
) +
# editar legenda para 1 linha
guides(fill = guide_legend(nrow = 1, byrow = TRUE)) +
# definir tema
theme_minimal() +
# editar tema
theme(
plot.title = element_text(hjust = 0.5, size = 16, face = "bold"),
plot.subtitle = element_text(hjust = 0.5),
legend.position = "top",
legend.title = element_blank(),
legend.justification = "center",
axis.text = element_text(size = 12),
axis.title = element_text(size = 12, face = "bold"),
plot.background = element_rect(fill = "white", color = "white")
) +
# inverter eixos
coord_flip()
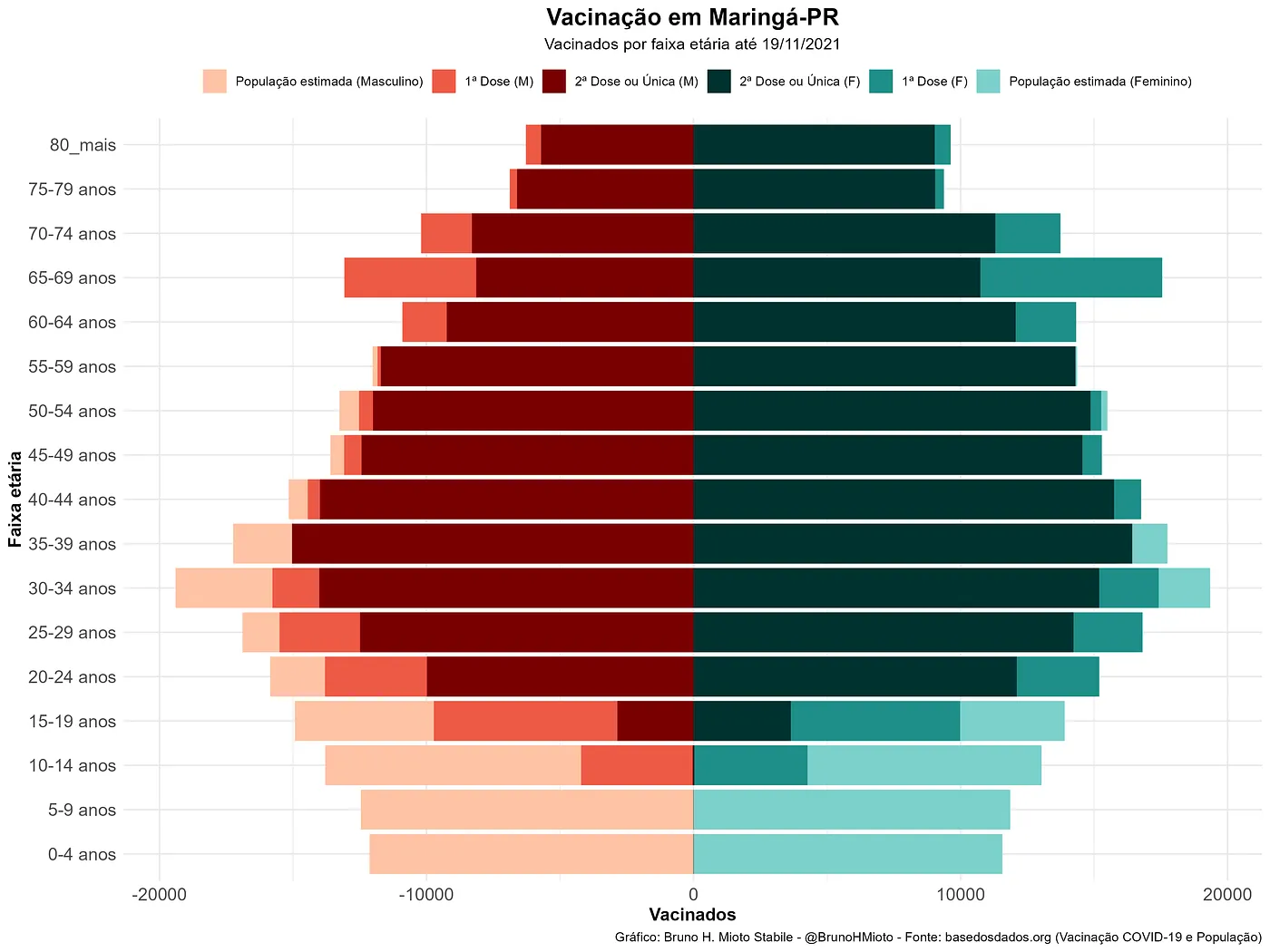
Gráfico completo
Ou então, podemos fazer um gráfico bem mais elaborado, com textos de apoio e várias outros detalhes. Perceba que neste gráfico adicionei alguns argumentos específicos para a população da minha cidade. Você pode editar para a sua também!
# definir o dataset e os eixos (invertidos)
ggplot(vacina_populacao2, aes(x = grupo_idade, y = n_grafico)) +
# populacao estimada
## feminino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
distinct(populacao_grafico, .keep_all = TRUE),
aes(
x = grupo_idade,
y = populacao_grafico,
fill = "População estimada (Feminino)"
),
stat = "identity"
) +
## masculino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
distinct(populacao_grafico, .keep_all = TRUE),
aes(
x = grupo_idade,
y = populacao_grafico,
fill = "População estimada (Masculino)"
),
stat = "identity"
) +
# 1_dose
## feminino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
filter(dose_vacina == "1"),
aes(fill = "1ª Dose (F)"),
stat = "identity"
) +
## masculino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
filter(dose_vacina == "1"),
aes(fill = "1ª Dose (M)"),
stat = "identity"
) +
# 2_dose
## feminino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
filter(dose_vacina == "2"),
aes(fill = "2ª Dose ou Única (F)"),
stat = "identity"
) +
## masculino
geom_bar(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
filter(dose_vacina == "2"),
aes(fill = "2ª Dose ou Única (M)"),
stat = "identity"
) +
# texto_1_dose
## feminino
geom_text(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
filter(dose_vacina == "1"),
# não colocar valores < 1%
aes(
x = grupo_idade, y = ifelse(porc < 1, NA, n_grafico),
label = ifelse(grupo_idade == "80_mais",
paste0(porc, "% do estimado (1ª Dose)"),
paste0(porc, "%")
),
hjust = 0
),
nudge_y = 100,
fontface = "bold"
) +
## masculino
geom_text(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
filter(dose_vacina == "1"),
# não colocar valores < 1%
aes(
x = grupo_idade, y = ifelse(porc < 1, NA, n_grafico),
label = ifelse(grupo_idade == "80_mais",
paste0(porc, "% do estimado (1ª Dose)"),
paste0(porc, "%")
),
hjust = 1
),
nudge_y = -100,
fontface = "bold"
) +
# texto_2_dose
## feminino
geom_text(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
filter(dose_vacina == "2"),
# não colocar valores < 1%
aes(
x = grupo_idade, y = ifelse(porc < 1, NA, n_grafico),
label = ifelse(grupo_idade == "80_mais",
paste0(porc, "% (2ª Dose)"),
paste0(porc, "%")
),
hjust = ifelse(porc < 50, 0, 1),
fontface = "bold"
),
# definir se aparecerá à esquerda ou à direita
nudge_y = ifelse(vacina_populacao2[vacina_populacao2$sexo %in% c("F"), ] %>%
filter(dose_vacina == "2") %>%
ungroup() %>%
select(porc) < 50, 100, -100),
color = "white"
) +
## masculino
geom_text(
data = vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
filter(dose_vacina == "2"),
# não colocar valores < 1%
aes(
x = grupo_idade, y = ifelse(porc < 1, NA, n_grafico),
label = ifelse(grupo_idade == "80_mais",
paste0(porc, "% (2ª Dose)"),
paste0(porc, "%")
),
hjust = ifelse(porc < 50, 1, 0),
fontface = "bold"
),
# definir se aparecerá à esquerda ou à direita
nudge_y = ifelse((vacina_populacao2[vacina_populacao2$sexo %in% c("M"), ] %>%
filter(dose_vacina == "2") %>%
ungroup() %>%
select(porc)) < 50, -100, 100),
color = "white"
) +
# texto estimativa
annotate("text",
x = "5-9 anos", y = -20000,
label = "População estimada pelo Ministério da Saúde (2020)",
fontface = "bold",
hjust = 0
) +
# seta estimativa
geom_curve(aes(x = 2.2, y = -17500, xend = "15-19 anos", yend = -15000),
size = 1.1, color = "#10002b",
curvature = -0.5,
arrow = arrow(length = unit(0.1, "in"))
) +
# texto feminino
annotate("text",
x = "0-4 anos", y = 5000,
label = "Feminino",
hjust = 0,
fontface = "bold"
) +
# texto masculino
annotate("text",
x = "0-4 anos", y = -5000,
label = "Masculino",
hjust = 1,
fontface = "bold"
) +
# eixos
labs(
x = "Faixa etária",
y = "Vacinados",
title = "Vacinação em Maringá-PR",
subtitle = "Vacinados por faixa etária até 19/11/2021",
caption = "Gráfico: Bruno H. Mioto Stabile - @BrunoHMioto - Fonte: basedosdados.org (Vacinação COVID-19 e População)"
) +
# definir as marcas no eixo y (futuramente eixo x)
scale_y_continuous(
breaks = c(-20000, -15000, -10000, -5000, 0, 5000, 10000, 15000, 20000),
labels = c(
"-20000" = "20.000",
"-15000" = "15.000",
"-10000" = "10.000",
"-5000" = "5.000",
"0" = "0",
"5000" = "5.000",
"10000" = "10.000",
"15000" = "15.000",
"20000" = "20.000"
)
) +
# renomear categoria do eixo x (futuramente eixo y)
scale_x_discrete(
labels = c(
"0-4 anos" = "0-4",
"5-9 anos" = "5-9",
"10-14 anos" = "10-14",
"15-19 anos" = "15-19",
"20-24 anos" = "20-24",
"25-29 anos" = "25-29",
"30-34 anos" = "30-34",
"35-39 anos" = "35-39",
"40-44 anos" = "40-44",
"45-49 anos" = "45-49",
"50-54 anos" = "50-54",
"55-59 anos" = "55-59",
"60-64 anos" = "60-64",
"65-69 anos" = "65-69",
"70-74 anos" = "70-74",
"75-79 anos" = "75-79",
"80_mais" = "80+"
),
position = "bottom"
) +
# definir as cores de acordo com o que colocamos no "fill" e no vetor "cores"
scale_fill_manual(
values = cores,
limits = c(
"População estimada (Masculino)",
"1ª Dose (M)",
"2ª Dose ou Única (M)",
"2ª Dose ou Única (F)",
"1ª Dose (F)",
"População estimada (Feminino)"
)
) +
# editar legenda para 1 linha
guides(fill = guide_legend(nrow = 1, byrow = TRUE)) +
# definir tema
theme_minimal() +
# editar tema
theme(
plot.title = element_text(hjust = 0.5, size = 16, face = "bold"), # título
plot.subtitle = element_text(hjust = 0.5), # subtítulo
legend.position = "top", # posição da legenda
legend.title = element_blank(), # retirar título da legenda
legend.justification = "center", # centralizar legenda
axis.text = element_text(size = 12), # editar texto dos eixos
axis.title = element_text(size = 12, face = "bold"), # editar títulos dos eixos
text = element_text(family = "Open Sans"), # utilizar a fonte Open Sans em todo o gráfico
plot.background = element_rect(fill = "white", color = "white") # fundo branco
) +
# inverter eixos x e y! (e definir limites laterais)
coord_flip(ylim = c(-20000, 20000))
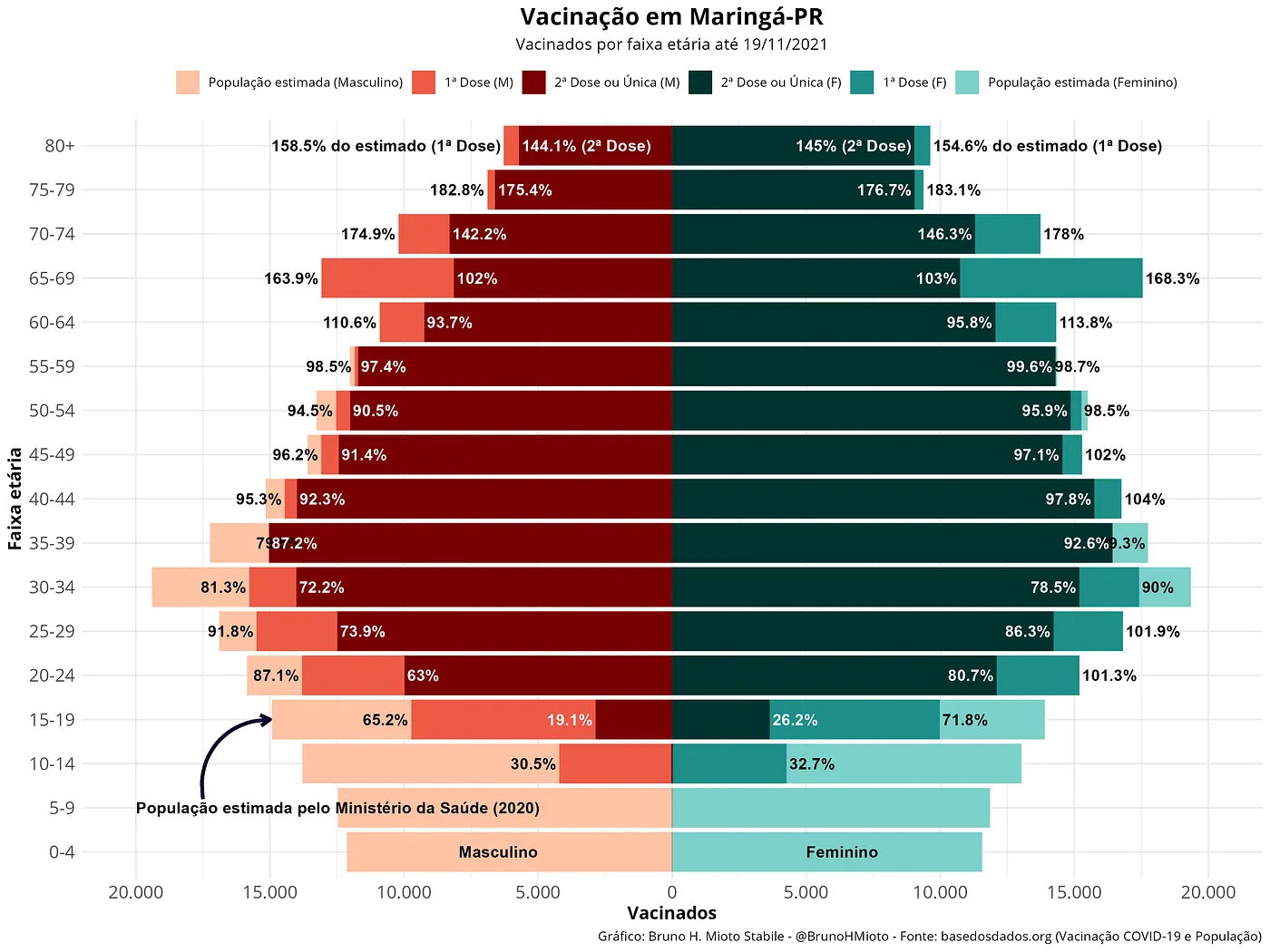
Algumas faixas etárias possuem bem mais que 100% de vacinados. Isso pode ser erro na projeçãom, na inserção dos dados no sistema, ou podem ter sidos vacinados idosos de outras cidades da região (Maringá é a cidade central na região), por exemplo. Estes são problemas que encontramos ao lidar com dados na pequena escala, como municípios. Ao utilizar dados para todo o país, muitos desses erros são compensados em larga escala.
Talvez você tenha percebido que na faixa etária de 35–39 anos os textos ficaram meio errados. Isso aconteceu por causa da aplicação de vacinas de dose única, que chegaram todas no mesmo período e foram aplicadas apenas nesse grupo da população. Dessa forma, o número de vacinados com 2ª dose + Dose Única é maior que os vacinados com a 1ª Dose. Claramente, quando fiz esse gráfico, não contava com esse detalhe, e talvez isso também ocorra na sua cidade. Fique a vontade para melhorar o código e compartilhar!
Notou algo errado ou tem uma sugestão?
Contribua com a BD editando este artigo via pull request no nosso GitHub.Base dos Dados